4月24日,山东大学微生物技术国家重点实验室佘群新教授团队在nature communications 杂志在线发表了题为“flexible tam requirement of tnpb enables efficient single-nucleotide editing with expanded targeting scope”的研究论文。佘群新教授、冯旭研究员为该论文的共同通讯作者,冯旭研究员与2021级博士生许如意为并列第一作者,山东大学微生物技术国家重点实验室为第一完成单位和通讯作者单位。
近几年来,一种由细菌is200/is605转座子元件编码的转座相关蛋白tnpb,被发现是一种新型的可编程rna引导核酸酶,其分子量仅400个氨基酸左右,远小于当前广泛使用的cas9和cas12编辑工具。研究表明tnpb能与转座子编码的小rna结合、形成核酸蛋白复合物,并在转座子邻近基序(tam)存在的情况下进行rna引导的特异性双链dna切割。然而,tnpb蛋白在微生物基因编辑中的特性与应用潜力亟待研究。
该研究系统解析了来自嗜热古菌的tnpb促进同源重组依赖的高效微生物基因组编辑的机制。通过对该tnpb的dna切割机制以及其在天然宿主中介导基因编辑特性的系统解析,他们发现该tnpb对于诱导细胞死亡和促进基因编辑有不同的tam需求。tnpb能够识别广泛的tam变体序列并介导基因编辑,包括众多不引起细胞死亡的tam。重要的是,tnpb对弱tam处的靶点表现出极高的靶向特异性,利用此特性,成功实现了基于tnpb的同源重组驱动的高效单核苷酸编辑。采用不同的弱tam序列有望克服当前限制crispr微生物基因编辑的两大瓶颈:转化效率低以及单核苷酸编辑难的问题。该微生物基因编辑新理论有望应用于多种crispr-cas系统。
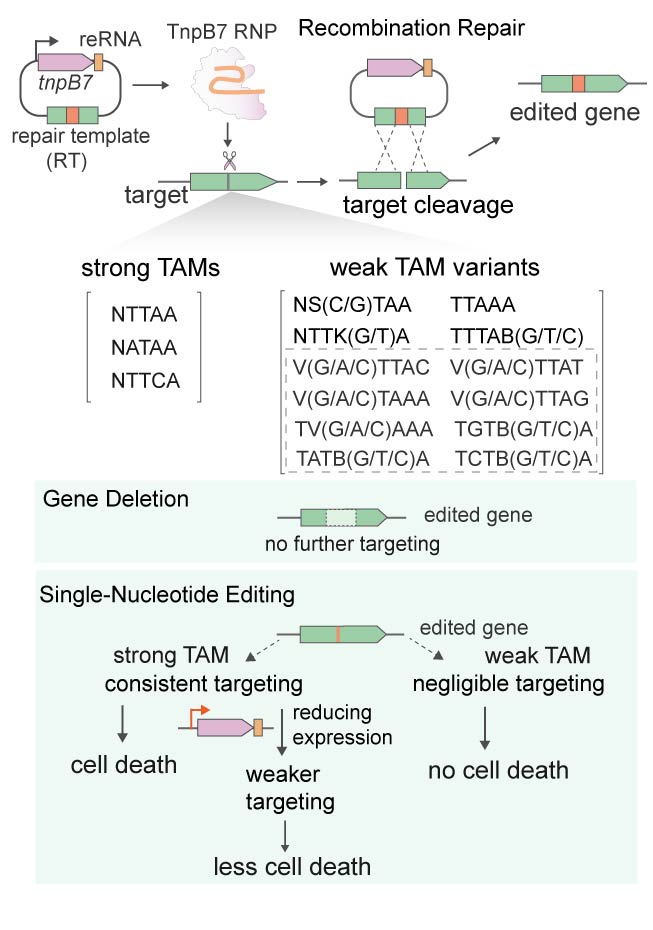
图1 tnpb灵活的tam需求促进了其在单核苷酸编辑中的应用
佘群新教授团队长期从事crispr与古菌生物学的研究,对iii型crispr系统的研究揭示了一系列相关的分子机制,并建立了基于其靶标rna切割活性的基因沉默技术;此外,他还率先发表了基于内源crispr系统的高效原核生物基因编辑技术。在nature、cell、molecular cell、cell host microbe、nucleic acids research、pnas、cell reports 和mbio 等著名科研杂志发表研究论文150余篇;近年来又开展了tnpb rna引导核酸酶和细菌的防御与抗防御机制的研究。本次在nature communications上发表的对古菌tnpb的研究成果是该团队的又一个重要进展。
中国科学院南海海洋科学研究所王晓雪教授团队和华中农业大学韩文元教授参与了该项目的部分研究工作。本项研究得到了科技部重点研发计划、国家自然科学基金、山东大学齐鲁青年启动基金、山东省科技厅基金、山东省教育厅基金以及微生物国家重点实验室揭榜挂帅等项目的资助。
原文链接:


