近日,海洋研究院海洋碳汇团队在advanced science杂志上发表题为“selective rna processing and stabilization are multi-layer and stoichiometric regulators of gene expression in escherichia coli”的研究论文。海洋研究院副研究员黄冉冉为论文通讯作者,药学院助理研究员刘代喜、海洋研究院硕士研究生吕海波、王亚菲为论文的共同第一作者。
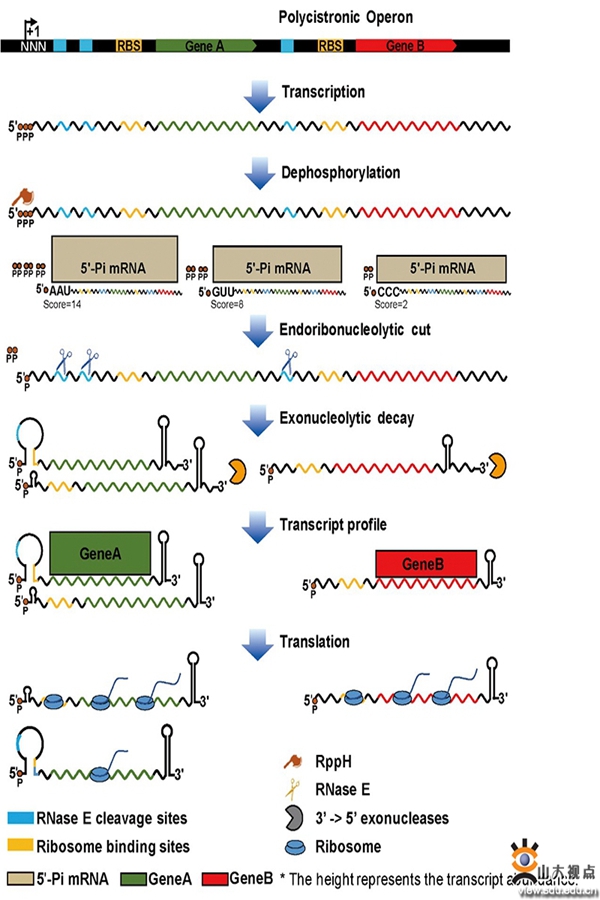
选择性rna剪切与保护机制(selective rna processing and stabilization, srps)是微生物调节蛋白复合体亚基化学计量数的重要转录后调控机制,能够调控多顺反子操纵子中多个基因的差异表达。然而,srps相关酶如何协同进行化学计量调节尚不清楚。
本研究通过rna末端测序以及比较转录组分析,首次在单核苷酸水平上对大肠杆菌srps相关酶全基因组范围内的作用靶点进行解析,揭示了焦磷酸水解酶(rpph)、rna内切酶(rnase e)和rna外切酶在决定基因转录丰度中的协同效应,解析了大肠杆菌srps的分子机制。具体来说,在转录后层面:转录本5'核苷酸序列决定其被rpph去除焦磷酸的比例,进而决定其被rna内切酶的切割效率,以及后续转录本中形成的二级结构的稳定程度决定rna外切酶在此处的终止效率,进而决定大肠杆菌中各转录本的丰度;同时,在翻译层面:转录本5'utr区不同rna内切酶的酶切位点导致5'utr形成不同二级结构,影响转录本上rbs元件与核糖体的结合,进而导致翻译效率的变化。最终,通过这两个层面的精细调控,定量调节操纵子内基因编码蛋白丰度的变化。同时,本研究使用双荧光报告系统验证了srps元件调控的有效性,并为进一步探索和利用srps元件构建了第一个原核srps位点数据库(prosrpsite,http://prosrpsite.sdu.edu.cn/)。
本研究揭示了大肠杆菌srps的分子机制,并确定了多层次、可定量和全局的调控机制。srps元件的可定量调控性使其可用于精确优化合成生物学中靶基因的化学计量表达,以解决合成生物学中“底盘细胞中多个基因的平衡表达”的问题。本研究具有极大的理论意义和应用价值,将为合成生物学的发展提供相应的理论储备和技术保障,有望推动合成生物学从定性、描述性、局部性研究向定量、理论化和系统化研究的变革。相关研究工作得到了国家自然科学基金、国家重点研发计划、山东省重点研发计划等科研项目的资助。
黄冉冉副研究员长期从事基于新一代测序数据的微生物生物信息学分析工作,一方面利用(宏)基因组、(宏)转录组测序等手段,鉴定微生物的代谢过程、调控方式,解析微生物的群落结构变化及变化的推动机制;另一方面通过单细胞等技术针对所解析的调控机制开展定量合成生物学应用研究。其系列研究成果以第一或通讯作者在nature communications、advanced science、chemical engineering journal、microbiology spectrum、biotechnology for biofuels等学术期刊上发表。
论文链接:


