近日,山东大学微生物技术国家重点实验室(研究院)张正研究员、李越中教授团队在nucleic acids research杂志(jcr 1区,5-year if = 16.4)发表了题为“gdpf: a data resource for the distribution of prokaryotic protein families across the global biosphere”的研究论文。微生物技术国家重点实验室博士后潘卓和海洋研究院助理研究员李单单为并列第一作者,微生物技术国家重点实验室李越中教授和张正研究员为共同通讯作者,山东大学为唯一作者单位。
微生物在地球上无处不在,隐藏在人们的皮肤、肠道和土壤、河流、海洋、空气等环境中,是影响人类健康、全球生态变化的关键因素。传统的微生物生物地理学研究侧重于揭示微生物在复杂环境中的系统发育多样性,而真正驱动相关生态过程的则是这些微生物所表现出的功能。因此,了解环境中微生物功能特性的分布将有助于从功能机理上理解微生物群落和自然地理之间的关系。近年来,大量研究已在特定环境中(例如人类肠道)基于家族量化了微生物群落功能,但尚缺乏全球生物圈微生物功能家族分布的全面数据。
作者先前的研究表明,全球范围微生物群落的基因组测序比例已达较高水平(microbiome,8:134)。在此基础上,本研究结合地球微生物组计划样本和已测序基因组信息,整合kegg、cdd、pfam、cog等功能家族分类注释系统,构建了原核生物功能家族全球分布数据库gdpf()。gdpf提供了36334个家族、19734个超家族、12089个kegg直系同源系的全球分布信息,涵盖了土壤、海洋、动物、植物、沉积物等典型环境。用户可以在数据库中浏览、搜索、下载每个条目在全球10000个微生物群落中的分布数据,并支持比较分析多个条目在不同环境间的分布差异。gdpf数据资源有助于揭示全球水平上微生物功能的地理分布模式、关键影响因素和宏观生态学规律,促进相关地球生态和人类健康研究。

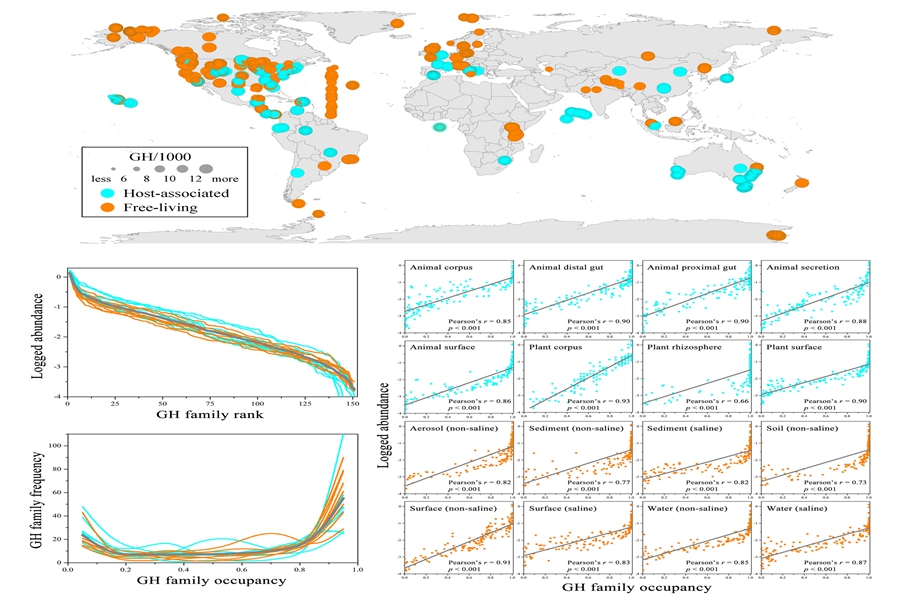
温室气体引起的全球变暖威胁着生物多样性、农业生产力和粮食安全。微生物编码的糖苷水解酶(gh)对碳水化合物的降解是全球碳循环的关键步骤。本研究全面调查了原核生物编码gh基因的全球生物地理分布模式,提供了不同环境条件下群落碳水化合物降解功能潜力的全景图。原核生物群落编码的gh组成存在明显的栖息地特异性,这种特异性是由多种生物因素和非生物因素共同驱动、通过高度确定性的组装过程产生的。该成果以“deterministic assembly processes shaping habitat-specific glycoside hydrolase composition”为题发表于global ecology and biogeography杂志(中科院1区,5-year if = 7.4),李单单助理研究员和张正研究员为本文并列第一作者,张正研究员和李越中教授为本文共同通讯作者,山东大学为唯一作者单位。
近年来,张正研究员、李越中教授团队在微生物群落生态领域取得了一系列研究成果,先后发表于nucleic acids research(2023和2022)、global ecology and biogeography(2023)、msystems(2023、2021和2020)、environmental microbiology(2023和2021)、microbiology spectrum(2021)、applied and environmental microbiology(2021)、computational and structural biotechnology journal(2021和2020)、microbiome(2020)等国际知名期刊。相关研究得到国家重点研发计划、国家科技基础资源调查专项、国家自然科学基金、中国博士后科学基金、山东省自然科学基金、山东大学齐鲁青年学者等项目资助。


