近日,微生物技术国家重点实验室霍刘杰教授、武大雷教授课题组以及医学融合与实践中心闫杰教授课题组合作,采用异源生物合成策略,在黏细菌中发现并表征了一个新颖的羊毛硫肽生物合成基因簇mfu,成功获得并表征了其产物myxococins的化学结构与抗炎生物活性,解析了其新颖的生物合成机制。研究成果以“discovery and characterization of a myxobacterial lanthipeptide with unique biosynthetic features and anti-inflammatory activity”为题在线发表于化学顶级期刊美国化学会会志journal of the american chemical society(中科院一区,if=16)。微生物技术国家重点实验室博士研究生王晓彤和博士生陈晓宇为论文第一作者,微生物技术国家重点实验室霍刘杰教授、武大雷教授与医学融合与实践中心闫杰教授为通讯作者。山东大学为第一完成单位。
黏细菌为土壤来源的革兰氏阴性细菌,可产生丰富的天然产物,具有巨大的生物合成潜力。生物信息学分析显示,黏细菌基因组中存在许多核糖体肽类生物合成基因簇,但目前仅有少量黏细菌来源的核糖体肽类化合物被报道,因此黏细菌来源的核糖体肽类天然产物具有极大的挖掘潜力。羊毛硫肽类化合物是目前为止研究最为广泛的一类核糖体肽,具有多样的结构和生物活性。本研究通过生物信息学分析,在黏细菌myxococcus fulvus mcy8288的基因组中发现了一个假定的羊毛硫肽生物合成基因簇mfu,其同时编码了ⅱ型羊毛硫肽合成酶mfum以及ⅰ型羊毛硫肽环化酶mfuc,以及包含c39蛋白酶结构域的转运蛋白mfut。值得注意的是,该基因簇还包含了一个未在核糖体肽类天然产物中表征过的m61家族肽酶mfup(图1)。采用在大肠杆菌中前体肽与其共表达的策略,结果表明前体肽首先被mfum 的脱水结构域催化5个连续的苏氨酸残基的脱水,形成dhb;紧接着 mfum的环化结构域发挥活性,完成 cys18、 cys20 以及 cys31 三个位点的环化;之后mfuc 负责了剩余两个位点 cys33 和 cys35 的环化,形成复杂、交联的五环结构。这是首次发现ⅰ型与ⅱ型羊毛硫肽生物合成酶共同参与产物生成硫醚环的现象(图1)。通过marfey、串联质谱及核磁联用,确定了脱水环化及前导肽切割后产物myxococin a的化学结构(图1)。
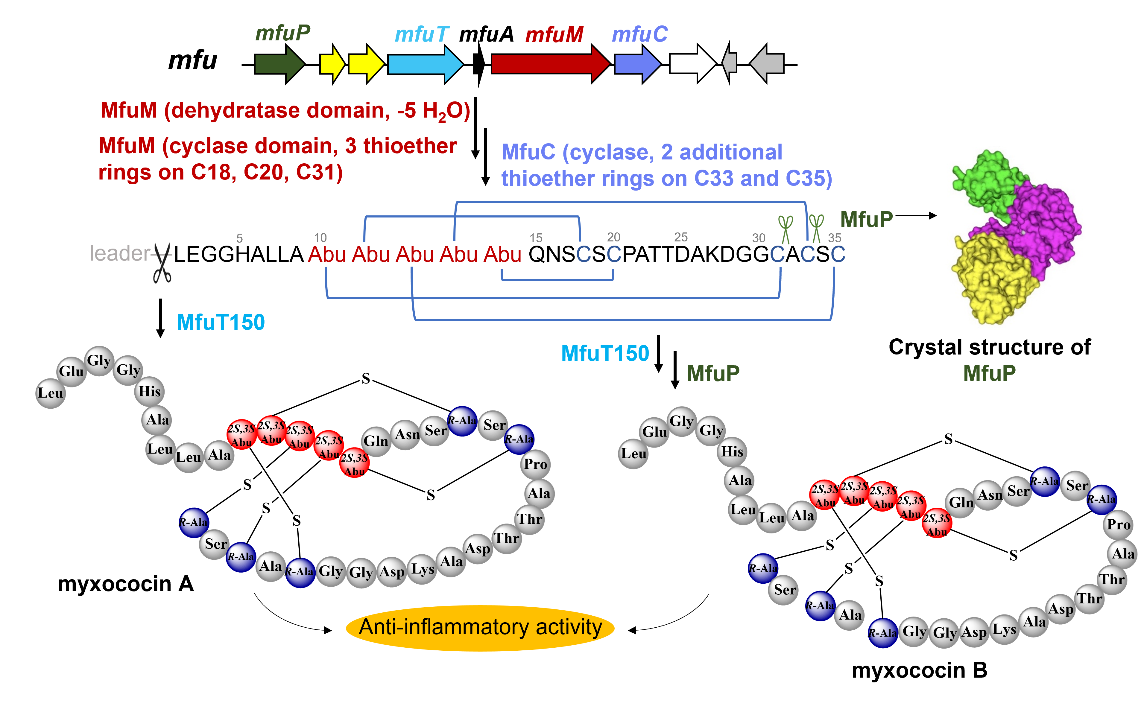
图1 myxococin的生物合成途径解析与化学结构表征
随后,通过体外生化活性表征,确定了m61家族肽酶mfup以严格的五环结构依赖的方式水解 myxococin a 中ala32与ser34 n端的两个酰胺键,发挥“解环”作用生成产物 myxococin b(图1)。这是首次报道该类“解环”翻译后修饰过程发生在核糖体肽类天然产物的生物合成过程中,也是m61家族肽酶首次被发现参与核糖体肽的生物合成。基于其新颖性,作者进一步对mfup进行了晶体结构的解析,并结合突变体工程确定了其活性位点(图2a-c)。结果显示mfup与已知结构的m61家族氨肽酶iloapn的整体结构类似,但是mfup的底物结合槽腔体的体积远大于iloapn(图2d),这与mfup识别并结合myxococin a的复杂五环结构相对应。此外,本论文还通过建立mfup与底物的结合模型(图2f),探究了mfup假定的底物结合口袋以及mfup与底物的相互作用。通过生物信息学分析发现,该类蛋白普遍编码于假定的核糖体肽类生物合成基因簇周围,暗示该类蛋白具有较大的挖掘潜力。
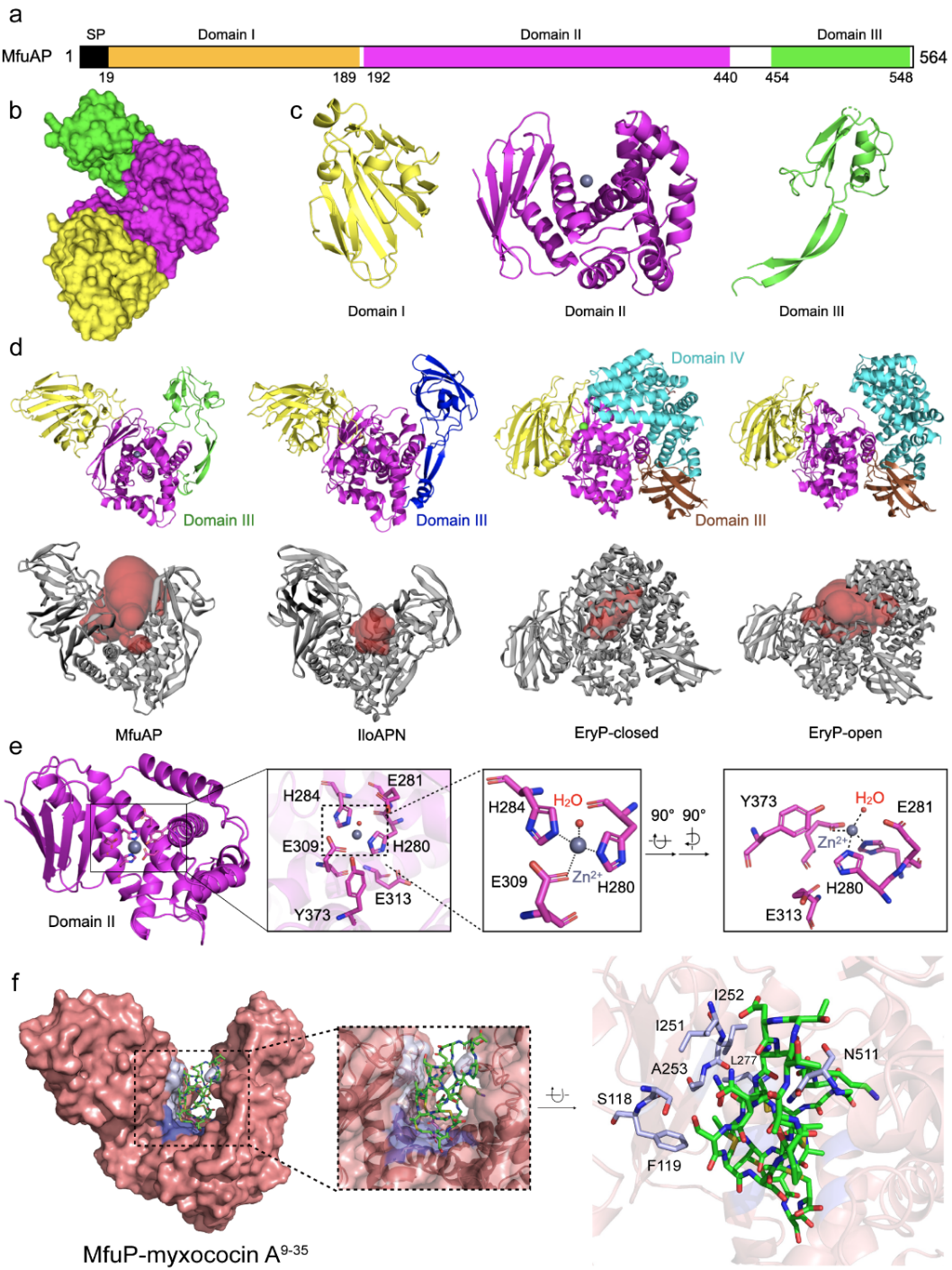
图2 mfup 的晶体结构解析。(a)mfup的一级结构示意图;(b)以surface模式展示的mfup整体结构;(c)mfup三个结构域的cartoon模型;(d)mfup、iloapn(pdb:4fgm)以及eryp封闭构象(pdb:7v9n)和开放构象(pdb:7v9q)的cartoon模型;(e)mfup活性中心锌离子与活性位点关键氨基酸的相互作用示意图;(f)mfup-myxococin a9-35的结合模式图
最后,作者对myxococin a和myxococin b进行了生物活性的检测,以lps诱导的小鼠单核巨噬细胞 raw264.7为模型,发现其对no的释放和诱导型no合酶(nos2),以及促炎细胞因子tnfα、il6和il1β在转录水平和蛋白水平上均具有明显的抑制效应,表明其良好的抗炎活性,且未检测到细胞毒性(图3)。该特性表明其具有进一步开发成为抗炎药物先导化合物的潜力,且其抗炎活性机制也值得进一步研究。
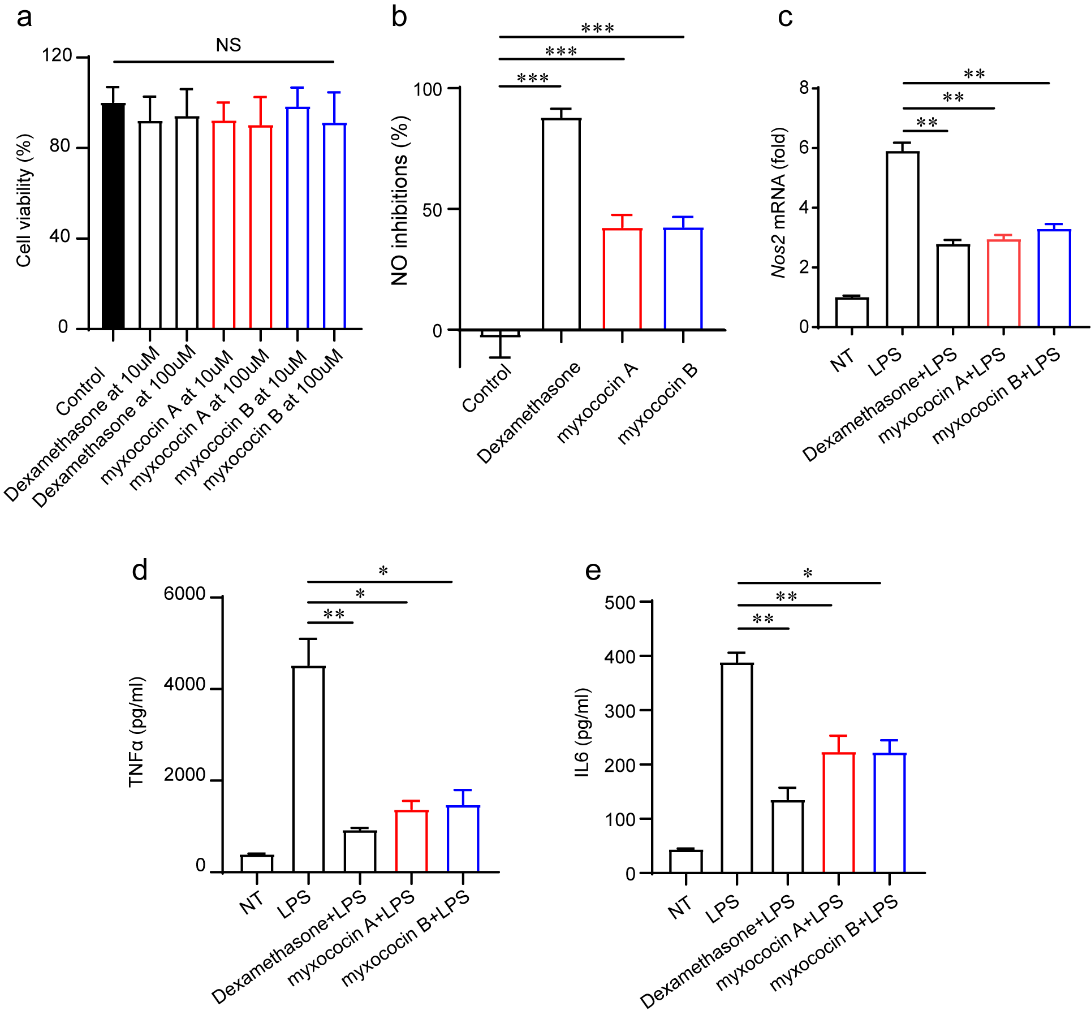
图3 myxococin a和b对raw264.7细胞株的细胞毒性和抗炎活性检测。阳性对照:地塞米松
该研究得到包括山东大学-亥姆霍兹国际实验室、国家重点研发计划,国家自然科学基金与山东省自然科学基金等项目资助。山东大学生命环境研究公共技术平台为本工作提供了重要支持。
论文链接:


